Table of Contents
Whole slide images en virtuele coupes
PMA.core ondersteunt een breed scala aan technieken en beeldmodaliteiten.
Een volledige lijst met ondersteunde bestandsformaten is beschikbaar op onze website via https://www.pathomation.com/formats.
U kunt meer lezen over onze filosofie omtrent ondersteuning van leverancier-specifieke bestandsindelingen in ons persbericht.
Een introductie waarin wordt ingegaan op problemen omtrent bestandsindelingen in digitale pathologie is beschikbaar via de Digital Pathology Today podcast.
Brightfield
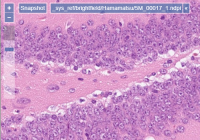
De PMA.core engine voor het inlezen van WSIs ondersteunt meer dan 35 verschillende, leverancier-specifieke bestandsindelingen die worden gebruikt om brightfield microscopie data vast te leggen. We bieden een mix van open source community indelingen (OMERO TIFF & ZARR, DICOM, …), maar ook legacy versies zoals Olympus Webview.
Er is een volledige, up-to-date lijst beschikbaar op onze website via https://www.pathomation.com/formats.
Fluorescentie
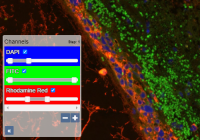
DAPI, Cy3, Cy5, … Als deze termen bekend klinken, is er goede kans dat u te maken hebt met fluorescentiemicroscopie. Wij hebben jarenlang met vele leveranciers gewerkt om dit type afbeelding optimaal te ondersteunen. U kunt , direct vanuit PMA.core individuele kanalen aan- en uitzetten, en histogrammen naar believen clippen.
Geavanceerde downstream applicaties (die bovenop PMA.core werken), zoals PMA.studio, kunnen zelfs het histogram van elk kanaal voor u visualiseren, alsmede gammacorrecties toepassen per individueel kanaal. Er is een volledige, up-to-date lijst beschikbaar van de ondersteunde fluorescentie bestandsindelingen op onze website via https://www.pathomation.com/formats.
Z-stacking
Zowel brightfield- als fluorescentiebeelden worden vaak gegenereerd op een microtoom, wat resulteert in gladde en platte coupes. Voor cytologische stalen, of wanneer microscopie wordt uitgevoerd bij het onderzoeken van gewassen, dan is z-stacking inmiddels de standaard geworden.
Uitstrijkjes en vergelijkbare materialen worden normaliter opgeslagen via Z-stacking,waarbij een staal wordt opgedeeld in minuscule stappen van één of twee microns per keer. Er is een volledige, up-to-date lijst beschikbaar van de ondersteunde Z-stacking bestandsindelingen op onze website via https://www.pathomation.com/formats.
Tijdserie
Stel dat u de celsamenstelling of embryo-ontwikkeling 24 uur lang wilt observeren. Sommige ontwikkelaars stellen u in staat om alle data van die tijdsperiode vast te leggen op één plek (mogelijkerwijs in combinatie met andere dimensies, zoals hierboven uitgelegd).
We hebben recent ondersteuning toegevoegd voor zulk soort data in tijdseries. We beschikken echter nog niet over veel referentie datasets, dus indien u zelf cases hebt voor zulke datasets, laat ons dit alstublieft weten. Wij kunnen dan met u samenwerken om de meest optimale manier te vinden waarop wij deze datasets kunnen presenteren voor uw specifieke (onderzoeks)project.
Geannoteerde formaten
Omdat een beeld meer zegt dan duizend woorden, worden WSIs vaak geannoteerd. Hier zijn enkele voorbeelden:
- Een instructeur wil de aandacht van een student vestigen op een bepaald onderdeel van een dia door pijlen in verschillende kleuren te tekenen en deze mogelijk te labelen.
- Een CRO laat pathologen annotaties maken op dia’s voor specifieke weefselsoorten, zoals necrotisch weefsel of tumorweefsel. De pathologen doen dit in overeenstemming met een vooraf vastgesteld kleurenschema.
- Een farmaceutisch bedrijf hanteert verschillende soorten tools voor het analyseren van afbeeldingen (AI/ML/DL); elk van deze tools produceert annotatielagen die dienen te worden geïntegreerd voor verdere studies en als vergelijkingsmateriaal.
Pathomation’s PMA.core onderscheidt drie verschillende soorten annotaties:
Annotatietype Format voorbeelden
| Type Annotation type | Voorbeelden | Kan lezen | Kan schrijven |
|---|---|---|---|
| Native | MRXS, NDPI(A), SVS | Yes | No |
| PMA.core | WKT | Yes | Yes |
| Third-party | MLD, XML | Yes | No |
Meer informatie omtrent de verschillende soorten annotaties en hoe deze op het Pathomation platform worden afgehandeld, vindt u in ons blogartikel.
Formulieren en meta-data
Synoptische rapporten vormen een geweldige manier om informatie te structureren die voorheen grotendeels ongestructureerd was.
Bijvoorbeeld: in plaats van dat een patholoog een rapport schrijft waarin wordt gesteld dat het weefsel van de patiënt abnormale tekenen van snel vermenigvuldigende celgroepen vertoont, laat u deze patholoog een formulier invullen met een selectievakje voor “TumorWeefselAanwezig”.
PMA.core ondersteunt het vastleggen van dit soort data en de mogelijkheid om formulieren te genereren. Zie voor meer informatie de specifieke sectie op deze wiki over dit onderwerp.
PMA.core is natuurlijk niet de enige tool ter wereld die dit kan. Wij erkennen dit door verschillende opties te bieden om externe meta-data in PMA.core te integreren. Voor meer informatie over dat onderwerp, verwijzen wij u graag naar ons blogartikel.
Overige bestandsformaten
Microsoft Excel en CSV
In PMA.core wordt veel informatie weergegeven in de vorm van lijsten: gebruikers,WSIs, formulieren… Het maakt niet uit hoe uitgebreid wij deze lijstinterfaces maken, er gaat toch een moment komen waarop u denkt “als ik toch maar dit anders kon indelen zoals _dit_, of als ik dit simpelweg naar _die_ persoon kon sturen…
De meeste weergaven in PMA.core hebben om die reden in de rechterbovenhoek een knop om naar Excel te exporteren waarmee u de huidige data als lijst kunt exporteren. Probeer dit maar eens uit. Als u niet kunt vinden wat u zoekt, laat het ons weten, zodat wij dit in onze volgende versie kunnen implementeren (we maken geen beloftes; we gaan eerst evalueren of uw verzoek functionaliteit oplevert die voor al onze gebruikers nuttig kan zijn).
Ondersteunde Codecs voor WSI
Het woord Codec is een combinatie van de woorden “coder” en “decoder”. Een deel van beide woorden wordt gebruikt om het gangbare woord “Codec” te creëren. Denk hierbij dus aan zowel een codeerder als decodeerder. Het coderen en decoderen van afbeeldingen is vereist om pixel om te zetten in stukjes informatie die kunnen worden verzonden en vertaald.
PMA.core ondersteunt de volgende codecs en standaarden omtrent het versleutelen van data:
- Blosc
- BMP
- Deflate
- iSyntax
- JPEG
- JPEG2000
- JPEGXR
- lossless
- LZW
- PNG
- RLE
Verdere en meer up-to-date informatie over hoe dit van toepassing is op specifieke bestandsindelingen, kan worden gevonden op onze website.