Surveillance de l'utilisation du système
- États de l'utilisateur (glossary)
paramètres
Diagnostics
Rapports
Surveillance de l'utilisation du système
paramètres
Diagnostics
Rapports
PMA.core supporte une variété de techniques et de modalités de capture d'images.
Une liste complète des formats actuellement pris en charge est disponible sur notre site Web à l'adresse https://www.pathomation.com/formats.
Vous pouvez en savoir plus sur notre philosophie concernant la prise en charge des formats de fichiers spécifiques aux fournisseurs dans notre communiqué de presse.
Une introduction discutant de la question du format de fichier dans la pathologie numérique est disponible dans le podcast Digital Pathology Today.
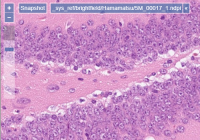
Le moteur de lecture de diapositives PMA.core prend en charge plus de 35 formats de fichiers différents, propres à chaque fournisseur, qui sont utilisés pour encapsuler les données de microscopie en champ clair. Nous proposons un mélange de formats communautaires open source (OMERO TIFF & ZARR, DICOM, …]), ainsi que des formats existants comme Olympus Webview.
Une liste complète et actualisée est disponible sur notre site Web à l'adresse https://www.pathomation.com/formats.
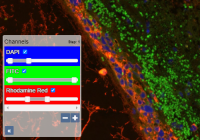
DAPI, Cy3, Cy5, … Si ces termes vous sont familiers, il y a de fortes chances pour que vous soyez engagé dans la microscopie. Nous avons travaillé avec de nombreux fournisseurs au fil des ans pour optimiser la prise en charge de ce type d'imagerie. Vous pouvez basculer les canaux individuels directement dans PMA.core, et faire des coupures d'histogramme comme vous le souhaitez. Les applications avancées en aval (qui s'exécutent au-dessus de PMA.core) comme PMA.studio peuvent même visualiser l'histogramme de chaque canal pour vous, et appliquer une correction gamma supplémentaire par canal.
Une liste actualisée des formats de fichiers fluorescents pris en charge est disponible sur notre site Web à l'adresse suivante https://www.pathomation.com/formats.
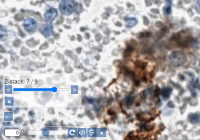
Les lames en champ clair et fluorescentes sont souvent générées sur un microtome, ce qui permet d'obtenir des surfaces lisses et plates. Cependant, lorsque l'on travaille avec des échantillons cytologiques, ou que l'on fait de la microscopie dans la recherche sur les cultures, ce n'est pas toujours le cas. Les frottis et autres matériaux similaires sont généralement capturés par zstacking, qui consiste à parcourir un échantillon par étapes minuscules d'un ou deux microns à la fois.
Une liste actualisée des formats de z-stacking pris en charge par PMA.core est disponible sur notre site Web à l'adresse suivante https://www.pathomation.com/formats.
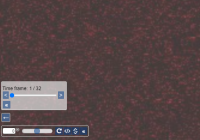
Supposons que vous vouliez observer une culture cellulaire ou le développement d'un embryon pendant une période de 24 heures. Certains fabricants permettent de capturer toutes les données de cette période de temps dans un seul conteneur (possible en combinaison avec d'autres dimensions comme celles mentionnées ci-dessus).
Récemment, nous avons ajouté le support pour ces données de séries temporelles. Nous n'avons pas encore beaucoup de jeux de données de référence, donc si vous avez des cas d'utilisation pour ces données, faites-le nous savoir. Nous pourrons alors travailler avec vous pour trouver la manière la plus optimale de les présenter afin de faire avancer votre projet (de recherche) spécifique.
Parce qu'une image en dit plus que mille mots, les diapositives sont souvent annotées. En voici quelques exemples :
PMA.core de Pathomation fait la distinction entre 3 types d'annotations :
| Type d'annotation | Exemples de format | Peut lire | Peut écrire |
|---|---|---|---|
| Natif | MRXS, NDPI(A), SVS | Oui | Non |
| PMA.core | WKT | Oui | Oui |
| tiers | MLD, XML | Oui | Non |
Plus d'informations sur les différents types d'annotations et la façon dont elles sont traitées dans la plateforme Pathomation se trouve dans notre article de blog.
Les rapports synoptiques constituent un excellent moyen de structurer des informations qui étaient auparavant largement non structurées.
Voici un exemple : au lieu de laisser un pathologiste rédiger un rapport dans lequel il indique que le tissu du patient montre des signes de prolifération anormale de groupes de cellules, vous lui faites remplir un formulaire avec une case à cocher pour “TumorTissuePresent”.
PMA.core supporte ce type de capture de données et la possibilité de générer des formulaires. Pour plus d'informations, voir la section spécifique sur le sujet dans ce wiki.
Bien sûr, PMA.core n'est pas le seul outil au monde capable de faire cela. Nous le reconnaissons en offrant diverses options pour intégrer des méta-données externes dans PMA.core également. Pour plus d'informations à ce sujet, nous vous renvoyons à notre article de blog.
 Dans PMA.core, beaucoup d'informations sont présentées sous forme de liste : utilisateurs,diapositives, formulaires…
Dans PMA.core, beaucoup d'informations sont présentées sous forme de liste : utilisateurs,diapositives, formulaires…
Peu importe l'étendue des interfaces de ces listes, il y aura toujours un moment où vous vous direz “si seulement je pouvais les reformater comme _ceci_, ou si je pouvais les envoyer à _cette_ dame…”.
La plupart des affichages dans PMA.core ont donc un bouton d'exportation Excel dans le coin supérieur droit qui vous permet d'exporter les données actuelles sous forme de liste. Allez-y, essayez, et si vous ne trouvez pas ce que vous cherchez, faites-le nous savoir pour que nous puissions l'incorporer dans la prochaine version (pas de promesses ; nous devrons encore évaluer dans quelle mesure votre demande serait une fonctionnalité utile pour l'ensemble de notre base d'utilisateurs).
Le mot Codec est une combinaison de deux mots : “codeur” et “décodeur”. Un morceau de chaque mot est utilisé pour créer le mot communément utilisé “Codec”. Pensez-y comme à un codeur et un décodeur.
Le codage et le décodage des images sont nécessaires pour convertir les pixels en bits d'informations et en ROIs qui peuvent être transmis et traduits.
PMA.core supporte actuellement les codecs et les normes de codage de données suivants :
Vous trouverez des informations supplémentaires et actualisées sur la manière dont cela s'applique à des formats de fichiers spécifiques sur notre website.
Voir notre section séparée.