Überwachung der Nutzung des Systems
- Benutzerzustände (glossary)
Einstellungen
Diagnose
Berichte
Überwachung der Nutzung des Systems
Einstellungen
Diagnose
Berichte
PMA.core unterstützt eine Vielzahl von Bildaufnahmetechniken und -modalitäten.
Eine vollständige Liste der derzeit unterstützten Formate finden Sie auf unserer Website unter https://www.pathomation.com/formats.
Mehr über unsere Philosophie bezüglich der Unterstützung von herstellerspezifischen Dateiformaten können Sie in unserer Pressemitteilung nachlesen.
Eine Einführung in die Problematik der Dateiformate in der digitalen Pathologie ist über den Podcast Digital Pathology Today verfügbar.
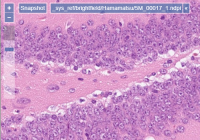
Die PMA.core slide reading engine unterstützt über 35 verschiedene herstellerspezifische Dateiformate, die zur Kapselung von Hellfeldmikroskopiedaten verwendet werden. Wir bieten eine Mischung aus Open-Source-Community-Formaten (OMERO TIFF & ZARR, DICOM, …) sowie älteren Formaten wie Olympus Webview.
Eine vollständige und aktuelle Liste finden Sie auf unserer Website unter https://www.pathomation.com/formats.
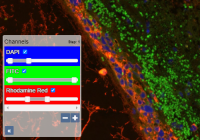
DAPI, Cy3, Cy5, … Wenn Ihnen diese Begriffe bekannt vorkommen, dann beschäftigen Sie sich mit Fluoreszenzmikroskopie. Wir haben im Laufe der Jahre mit vielen Anbietern zusammengearbeitet, um diese Art der Bildgebung optimal zu unterstützen. Sie können einzelne Kanäle direkt in PMA. core umschalten und das Histogramm nach Belieben beschneiden. Fortgeschrittene Downstream-Anwendungen (die auf PMA.core aufsetzen) wie PMA.studio können sogar das Histogramm jedes Kanals für Sie visualisieren und eine zusätzliche Gammakorrektur für jeden einzelnen Kanal vornehmen.
Eine aktuelle Liste der unterstützten Fluoreszenzdateiformate finden Sie auf unserer Website unter https://www.pathomation.com/formats.
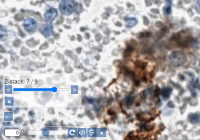
Sowohl Hellfeld- als auch Fluoreszenz-Objektträger werden häufig auf einem Mikrotom hergestellt, was zu glatten, ebenen Flächen führt. Bei der Arbeit mit zytologischen Proben oder bei der Mikroskopie in der Pflanzenforschung ist dies jedoch nicht immer der Fall. Ausstriche und ähnliche Materialien werden in der Regel durch Z-Stapelung erfasst, wobei eine Probe in winzigen Schritten von jeweils einem oder zwei Mikron durchlaufen wird.
Eine aktuelle Liste der von PMA.core unterstützten Z-Stacking-Formate finden Sie auf unserer Website unter https://www.pathomation.com/formats.
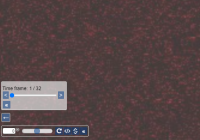
Nehmen wir an, Sie wollen eine Zellkultur oder die Entwicklung eines Embryos über einen Zeitraum von 24 Stunden beobachten. Einige Hersteller ermöglichen die Erfassung aller Daten aus diesem Zeitraum in einem einzigen Behälter (möglicherweise in Kombination mit anderen Dimensionen wie den oben genannten).
Vor kurzem haben wir die Unterstützung für diese Zeitreihendaten hinzugefügt. Wir haben allerdings noch nicht allzu viele Referenzdatensätze. Wenn Sie also Anwendungsfälle für diese Daten haben, lassen Sie es uns bitte wissen. Wir können dann mit Ihnen zusammenarbeiten, um die optimalste Art der Darstellung für Ihr spezifisches (Forschungs-)Projekt zu finden.
Da ein Bild mehr sagt als tausend Worte, werden Folien oft mit Anmerkungen versehen. Hier sind einige Beispiele:
PMA.core von Pathomation unterscheidet zwischen 3 verschiedenen Arten von Annotationen:
| Anmerkungsart | Formatbeispiele | Kann lesen | Kann schreiben |
|---|---|---|---|
| Einheimische | MRXS, NDPI(A), SVS | Ja | Nein |
| PMA.core | WKT | Ja | Ja |
| Drittanbieter | MLD, XML | Ja | Nein |
Weitere Informationen zu den verschiedenen Arten von Anmerkungen und wie sie in der Pathomation-Plattform gehandhabt werden, finden Sie in unserem Blog-Artikel.
Die synoptische Berichterstattung ist eine großartige Möglichkeit, Informationen zu strukturieren, die zuvor weitgehend unstrukturiert waren.
Ein Beispiel: Anstatt einen Pathologen einen Bericht schreiben zu lassen, in dem er feststellt, dass das Gewebe des Patienten Anzeichen für abnorm wuchernde Zellgruppen aufweist, lassen Sie ihn ein Formular mit einem Kontrollkästchen für “TumorTissuePresent” ausfüllen.
PMA.core unterstützt diese Art der Datenerfassung und die Möglichkeit, Formulare zu erstellen. Weitere Informationen finden Sie in dem entsprechenden Abschnitt in diesem Wiki.
Natürlich ist PMA.core nicht das einzige Tool auf der Welt, das dies tun kann. Wir tragen dem Rechnung, indem wir verschiedene Optionen anbieten, um auch externe Metadaten in PMA.core zu integrieren. Für weitere Informationen zu diesem Thema verweisen wir auf unseren Blog-Artikel.
In PMA.core werden viele Informationen in Form einer Liste dargestellt: Benutzer, Folien, Formulare…
Egal, wie umfangreich wir diese Listenschnittstellen gestalten, irgendwann werden Sie denken: “Wenn ich sie nur so umformatieren könnte, oder wenn ich sie einfach an _diese_ Dame schicken könnte…
Die meisten Ansichten in PMA.core haben daher eine Excel-Export-Schaltfläche in der oberen rechten Ecke, mit der Sie die aktuellen Daten als Liste exportieren können. Probieren Sie es aus, und wenn Sie nicht finden, wonach Sie suchen, lassen Sie es uns wissen, damit wir es in die nächste Version aufnehmen können (keine Versprechungen; wir müssen noch prüfen, inwieweit Ihr Wunsch eine nützliche Funktion für unsere gesamte Benutzerbasis ist).
Das Wort Codec ist eine Kombination aus den beiden Wörtern “coder” und “decoder”. Ein Teil von jedem Wort wird verwendet, um das allgemein verwendete Wort “Codec” zu bilden. Stellen Sie sich dies als Kodierer und Dekodierer vor. Die Kodierung und Dekodierung von Bildern ist erforderlich, um Pixel in Informationsbits und ROIs umzuwandeln, die übertragen und übersetzt werden können. PMA.core unterstützt derzeit die folgenden Codecs und Datenkodierungsstandards:
Weitere und aktuelle Informationen darüber, wie dies für bestimmte Dateiformate gilt, finden Sie auf unserer Website.
Siehe our separate section.